パッケージの概要
parsnipは、機械学習や予測モデリングを行うパッケージ群tidymodelsに含まれるパッケージで、様々なモデルを統一的な記法で使用するためのインターフェースを提供します。 一つのアルゴリズム、例えばランダムフォレストのモデルを構築しようとしたときに、パッケージはranger、randomForest等複数の選択肢が存在し、その実行方法やパラメータの名称、設定方法は微妙に異なっていることもあるかもしれません。 parsnipは、これらのパッケージ間の実行方法の違いを吸収してくれるパッケージです。
parsnipの概要を示す例として、ランダムフォレストによる回帰モデルを作成します。 サンプルデータとして、modeldataパッケージに含まれているカリフォルニア州サクラメント市の住宅価格のデータSacramentoを用います。
library (parsnip)library (modeldata)library (dplyr)set.seed (1234 )data ("Sacramento" , package= "modeldata" ) # 今回はカーディナリティの高い(レコード数に対して取り得る値が多い)特徴量は除外 <- Sacramento[, ! (colnames (Sacramento) %in% c ("city" , "zip" , "latitude" , "longitude" ))] str (d)
tibble [932 × 5] (S3: tbl_df/tbl/data.frame)
$ beds : int [1:932] 2 3 2 2 2 3 3 3 2 3 ...
$ baths: num [1:932] 1 1 1 1 1 1 2 1 2 2 ...
$ sqft : int [1:932] 836 1167 796 852 797 1122 1104 1177 941 1146 ...
$ type : Factor w/ 3 levels "Condo","Multi_Family",..: 3 3 3 3 3 1 3 3 1 3 ...
$ price: int [1:932] 59222 68212 68880 69307 81900 89921 90895 91002 94905 98937 ...
まずはrandomForestパッケージを使用します。 ここでは各パッケージをそのまま使用する場合とparsnipを使用する場合との違いを示すことが目的ですので詳細は割愛しますが、Sacramentoデータのpriceを目的変数として、他の変数で回帰するランダムフォレストモデルを構築しています。
# From randomForest library (randomForest)<- randomForest (~ ., data = d, mtry = 3 , ntree = 200 , nodesize = 3 ,importance = TRUE
Call:
randomForest(formula = price ~ ., data = d, mtry = 3, ntree = 200, nodesize = 3, importance = TRUE)
Type of random forest: regression
Number of trees: 200
No. of variables tried at each split: 3
Mean of squared residuals: 7646314367
% Var explained: 55.48
次にrangerパッケージで同じ内容のランダムフォレストモデルを構築します。 この二つのパッケージの例では実行方法に大きな違いはありませんが、それでもなお、指定するハイパーパラメータの変数名が一部異なっていることがわかります。
# From ranger library (ranger)<- ranger (~ ., data = d, mtry = 3 , num.trees = 200 , min.node.size = 3 ,importance = "impurity"
Ranger result
Call:
ranger(price ~ ., data = d, mtry = 3, num.trees = 200, min.node.size = 3, importance = "impurity")
Type: Regression
Number of trees: 200
Sample size: 932
Number of independent variables: 4
Mtry: 3
Target node size: 3
Variable importance mode: impurity
Splitrule: variance
OOB prediction error (MSE): 7674868879
R squared (OOB): 0.5536376
今度はparsnipを通して両パッケージのランダムフォレストを実行します。 二つの実行例のコードを見比べるとわかるように、parsnipを通して実行することで多くのパラメータが同じ変数名で指定できるようになります。
# From randomForest rand_forest (mtry = 3 , trees = 200 , min_n = 3 ) %>% set_mode ("regression" ) %>% set_engine ("randomForest" , importance = TRUE ) %>% fit (price ~ ., data = d)
parsnip model object
Call:
randomForest(x = maybe_data_frame(x), y = y, ntree = ~200, mtry = min_cols(~3, x), nodesize = min_rows(~3, x), importance = ~TRUE)
Type of random forest: regression
Number of trees: 200
No. of variables tried at each split: 3
Mean of squared residuals: 7633083891
% Var explained: 55.56
# From ranger rand_forest (mtry = 3 , trees = 200 , min_n = 3 ) %>% set_mode ("regression" ) %>% set_engine ("ranger" , importance = "impurity" ) %>% fit (price ~ ., data = d)
parsnip model object
Ranger result
Call:
ranger::ranger(x = maybe_data_frame(x), y = y, mtry = min_cols(~3, x), num.trees = ~200, min.node.size = min_rows(~3, x), importance = ~"impurity", num.threads = 1, verbose = FALSE, seed = sample.int(10^5, 1))
Type: Regression
Number of trees: 200
Sample size: 932
Number of independent variables: 4
Mtry: 3
Target node size: 3
Variable importance mode: impurity
Splitrule: variance
OOB prediction error (MSE): 7655884183
R squared (OOB): 0.5547417
この例のように、ランダムフォレストという同じモデルの同じハイパーパラメータであっても、パッケージによって変数名は異なっている場合があります。 また、パッケージによっては、目的変数と説明変数をformula形式(y ~ xのような形式)ではなく、それぞれを引数で与える方式を採用しているかもしれません。 parsnipはそのような差異を吸収し、統一的な記法でモデルを構築する仕組みを提供するパッケージです。
基本的な使用例1 - 回帰モデル
前項で示したランダムフォレストの例について、1ステップごとに解説しながら、parsnipの基本的な使用方法を紹介します。
(1) モデルの生成
ランダムフォレストの場合、モデル生成関数はrand_forest関数を使用します。 次のコードで、rand_forest関数にハイパーパラメータを与えて実行し、ランダムフォレストのmodel_specオブジェクトが作成されています。
<- rand_forest (mtry = 3 , trees = 200 )
Random Forest Model Specification (unknown mode)
Main Arguments:
mtry = 3
trees = 200
Computational engine: ranger
parsnipでは、モデルに与える引数は大きく、メイン引数(main arguments)とエンジン引数(engine arguments)に分けられます。 メイン引数は、ある特定のモデルにおいて、パッケージの種類によらずどのパッケージでも指定する必要があるような基本的なモデルのパラメータを指し、モデルの種類(およびその生成関数)によって決められています。
メイン引数は、モデルの生成関数を呼び出す際に引数として渡します。 上の例ではランダムフォレストの生成関数であるrand_forest関数に、メイン引数として二つの引数(一つ一つの決定木を作成する際に用いる特徴量の数”mtry”、および決定木の数”trees”)を渡しています。 この二つのパラメータは、パッケージによって変数名は異なる場合があるものの、ランダムフォレストを実行するためにはどのパッケージでも指定しなければならないような基本的なパラメータです。(省略した場合、デフォルトの値が設定されます。)
これに対してエンジン引数は、エンジンに指定したパッケージで使用できる引数のうち、メイン引数以外のものを指しています。 エンジン引数はモデルのパッケージ(エンジン)を指定する際に合わせて引数として指定するので、「エンジンの設定」の項で解説します。
(2) モードの設定
<- set_mode (rf_spec, mode = "regression" )
Random Forest Model Specification (regression)
Main Arguments:
mtry = 3
trees = 200
Computational engine: ranger
実行結果を見ると、1行目の(unknown)となっていた箇所が、(regression)に更新されたことが確認できます。
指定できるモードの種類はモデルの種類ごとに決まっています。 ランダムフォレストでは、回帰に用いる場合の”regression”以外にも、分類に用いる場合の”classification”および生存時間分析に用いる場合の“censored regression”が指定できます。 また、モデルの種類によっては、教師なし学習のモデルに用いる”clustering”、生存時間分析のモデルに用いる“risk regression”といったモードが用意されています。
(3) エンジンの設定
<- set_engine (rf_spec, engine = "randomForest" , importance = TRUE )
Random Forest Model Specification (regression)
Main Arguments:
mtry = 3
trees = 200
Engine-Specific Arguments:
importance = TRUE
Computational engine: randomForest
実行結果を見ると、最終行に表示されているエンジンの指定が、デフォルトの”ranger”から”randomForest”に更新されたことが確認できます。
上の例では、set_engine関数に”importance”という引数も渡しています。 このimportance引数は、randomForestパッケージでランダムフォレストを実行する際に使用する引数で、特徴量重要度を算出するかどうかを制御するためのものです。 randomForestパッケージで使用されている引数でありながら、parsnipのrand_forest関数ではメイン引数となっていない引数なので、先に述べた「エンジン引数」に該当します。 このようにエンジン引数は、使用するエンジンの指定と併せて、エンジンとして使用するパッケージ内での引数名のまま、set_engine関数に引き渡して使用することができます。
(4) モデルのフィッティング
# train dataとtest dataに分割 <- floor (nrow (d) * 0.75 )<- d[1 : n_train,]<- d[(n_train+ 1 ): nrow (d),]# モデルのフィッティング <- fit (rf_spec, price ~ ., data = d_train)
parsnip model object
Call:
randomForest(x = maybe_data_frame(x), y = y, ntree = ~200, mtry = min_cols(~3, x), importance = ~TRUE)
Type of random forest: regression
Number of trees: 200
No. of variables tried at each split: 3
Mean of squared residuals: 7699846058
% Var explained: 51.66
なお、説明変数と目的変数の関係をformula形式で指定する方法以外にも、説明変数と目的変数をそれぞれ引数xとyで直接指定する方式のfit_xy関数が用意されています。
<- fit_xy (rf_spec, x = d_train[, names (d_train)[names (d_train) != "price" ]], y = d_train$ price)
parsnip model object
Call:
randomForest(x = maybe_data_frame(x), y = y, ntree = ~200, mtry = min_cols(~3, x), importance = ~TRUE)
Type of random forest: regression
Number of trees: 200
No. of variables tried at each split: 3
Mean of squared residuals: 7671604694
% Var explained: 51.83
さて、(1)から(4)では解説のために、モデルの生成から学習までの一連の処理を、一つ一つのステップへと分解して実行してきました。 実際に使用する際は、以下のようにパイプ演算子でつなぎながら実行することで流れがわかりやすくなります。
<- rand_forest (mtry = 3 , trees = 200 ) %>% set_mode (mode = "regression" ) %>% set_engine (engine = "randomForest" , importance = TRUE ) %>% fit (price ~ ., data = d_train)
(補足) モデルスペックの様々な設定方法
まずは、モデルのモードとエンジンはそれぞれset_mode関数およびset_engine関数を使用して設定しましたが、モデル生成関数の引数に指定して設定することもできます。
# モデル生成用の関数(この例ではrand_forest)でモードとエンジンを設定する方式 <- rand_forest (mode = "regression" ,engine = "ranger" ,mtry = 3 , trees = 200
続いて、一度作成したmodel_specのメイン引数(ハイパーパラメータ)を更新するupdate関数を紹介します。 次のコードは、上の例で作成したランダムフォレストのmodel_specについて、mtryの更新および設定していなかったmin_nを新たに設定しています。
<- update (rf_spec, mtry = 5 , min_n = 3 , fresh = FALSE ) # fresh=TRUEにするとパラメータ全体を入れ替える # (この場合、update関数で指定していないtreesパラメータの設定は初期化される)
最後に、model_specのモードとパラメータを維持したまま他のエンジンに変換する、translate関数を紹介します。 次の例では、上で生成したランダムフォレストのmodel_specを、rangerエンジンからrandomForestエンジンに変換しています。 なお、エンジン引数を設定している場合、エンジン引数もそのまま引き継がれますが、前述のようにエンジン引数は本来的にパッケージごとに異なるパラメータですので、変換後のパッケージでは使用できないこともある点に注意が必要です。
translate (rf_spec, engine = "randomForest" )
Random Forest Model Specification (regression)
Main Arguments:
mtry = 5
trees = 200
min_n = 3
Computational engine: randomForest
Model fit template:
randomForest::randomForest(x = missing_arg(), y = missing_arg(),
mtry = min_cols(~5, x), ntree = 200, nodesize = min_rows(~3,
x))
(5) モデルによる予測
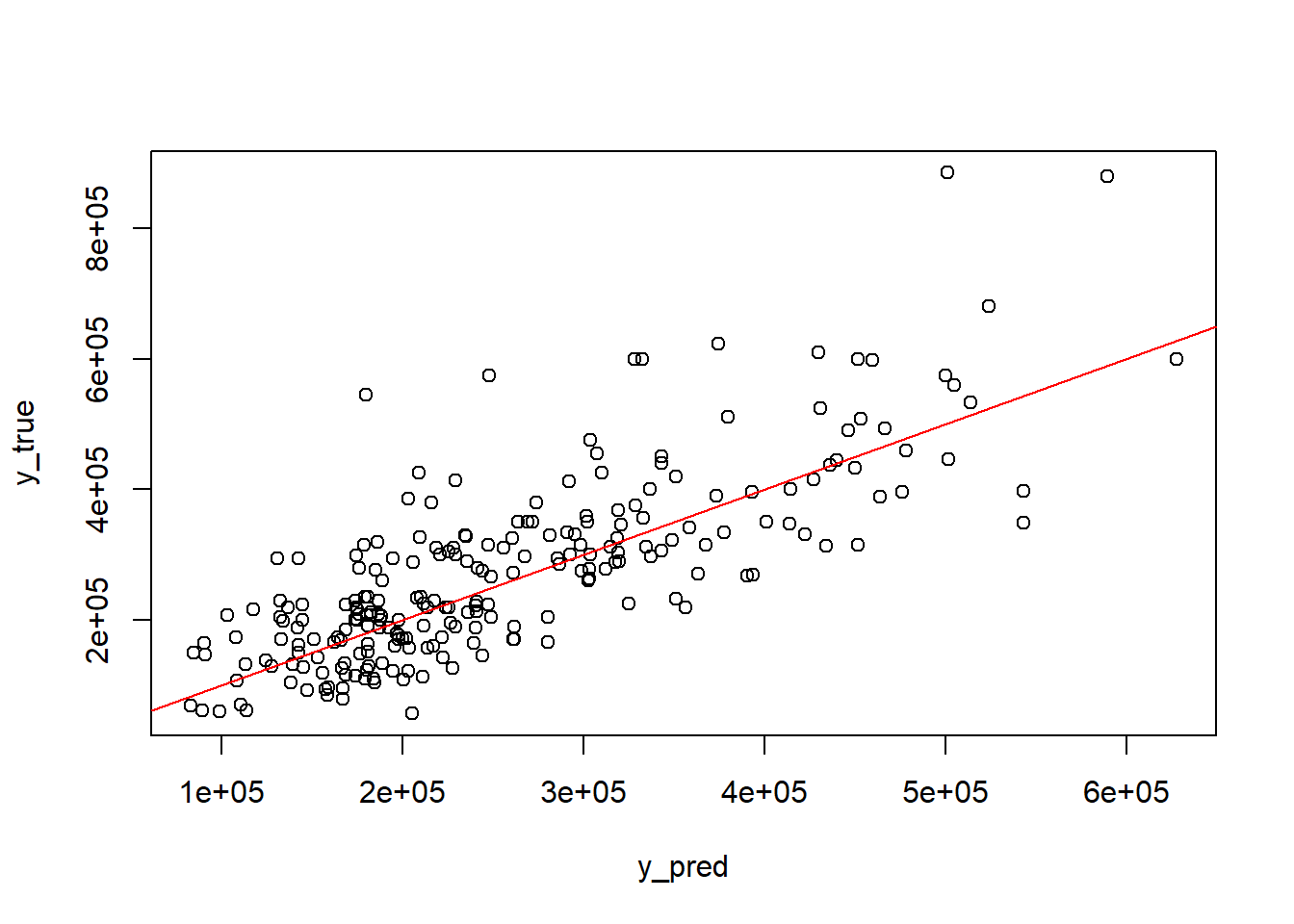
= predict (rf_fit, d_test)[[1 ]]= d_test$ priceplot (x = y_pred, y = y_true)abline (a= 0 , b= 1 , col= "red" )
なお予測値を取得する関数として、predict関数以外にも、parsnipではaugment関数が用意されています。 augment関数は学習済のモデルと予測対象のデータを引数にとり、データに対して予測値と残差のカラムを追加したデータセットを返します。
augment (rf_fit, d_test)[1 : 10 ,]
# A tibble: 10 × 7
.pred .resid beds baths sqft type price
<dbl> <dbl> <int> <dbl> <int> <fct> <int>
1 189186. 70814. 3 2 1196 Residential 260000
2 302809. -42809. 3 2 1621 Residential 260000
3 303272. -39772. 3 2 1811 Residential 263500
4 249206. 17304. 3 2 1540 Residential 266510
5 390607. -122857. 4 2.5 2647 Residential 267750
6 393610. -123610. 2 2 2750 Residential 270000
7 363463. -92463. 3 2.5 1910 Residential 271000
8 261279. 11421. 4 2.5 1846 Residential 272700
9 244198. 30802. 3 2 1543 Residential 275000
10 299032. -23032. 5 3 2494 Residential 276000
基本的な使用例2 - 分類モデル
ここまでは、ランダムフォレストの回帰モデルを構築する例を通して、parsnipの基本的な使用方法を解説してきました。 本項では、parsnipの別の使用例として、勾配ブースティング決定木(GBM)を分類問題に適用する例を紹介します。
この使用例を通じて、モデルの種類やモードが異なる場合でも、parsnipでは同じ流れでモデル構築ができることを示します。 また、学習済のparsnipモデルから情報を取り出して、更なる分析に使用する方法を紹介します。
サンプルデータとして、modeldataパッケージに含まれるcredit_dataを使用します。 顧客の信用度を示すStatus変数について、goodとbadを分類します。
<- na.omit (credit_data)# factor型の説明変数は数値に変換(ラベルエンコーディング)する。 # ※factor型のままでも自動的にone-hotエンコーディングされるためモデルの構築は可能。 # 今回は後続の説明上の理由からラベルエンコーディングしている。 <- c ('Home' , 'Marital' , 'Records' , 'Job' )<- (sapply (d2[, fct_vars], as.integer))str (d2)
'data.frame': 4039 obs. of 14 variables:
$ Status : Factor w/ 2 levels "bad","good": 2 2 1 2 2 2 2 2 2 1 ...
$ Seniority: int 9 17 10 0 0 1 29 9 0 0 ...
$ Home : int 6 6 3 6 6 3 3 4 3 4 ...
$ Time : int 60 60 36 60 36 60 60 12 60 48 ...
$ Age : int 30 58 46 24 26 36 44 27 32 41 ...
$ Marital : int 2 5 2 4 4 2 2 4 2 2 ...
$ Records : int 1 1 2 1 1 1 1 1 1 1 ...
$ Job : int 2 1 2 1 1 1 1 1 2 4 ...
$ Expenses : int 73 48 90 63 46 75 75 35 90 90 ...
$ Income : int 129 131 200 182 107 214 125 80 107 80 ...
$ Assets : int 0 0 3000 2500 0 3500 10000 0 15000 0 ...
$ Debt : int 0 0 0 0 0 0 0 0 0 0 ...
$ Amount : int 800 1000 2000 900 310 650 1600 200 1200 1200 ...
$ Price : int 846 1658 2985 1325 910 1645 1800 1093 1957 1468 ...
- attr(*, "na.action")= 'omit' Named int [1:415] 30 114 144 153 158 177 195 206 240 241 ...
..- attr(*, "names")= chr [1:415] "30" "114" "144" "153" ...
(1) モデルの生成~学習
# train dataとtest dataに分割 <- floor (nrow (d2) * 0.75 )<- d2[1 : n_train,]<- d2[(n_train+ 1 ): nrow (d2),]# モデルの生成~フィッティング <- boost_tree (mtry = 0.8 , # 木ごとに特徴量をサンプリングする割合 trees = 2000 , # 作成する木の本数 min_n = 1 , # 葉を分岐するために必要な最小のサンプル数 tree_depth = 5 , # 木ごとの最大の深さ learn_rate = 0.05 , # 学習率 sample_size = 0.8 , # 木ごとにデータをサンプリングする割合 stop_iter = 300 # アーリーストッピング(一定のラウンド数で精度が上がらなければ打ち切る)を判定するラウンド数 %>% set_mode (mode = "classification" ) %>% set_engine (engine = "xgboost" , eval_metric = "logloss" , # 損失関数の種類を指定 counts = FALSE , # mtryを割合で指定するオプション validation = 0.25 ) %>% # アーリーストッピングの判定に用いるデータの割合 fit (Status ~ ., data = d_train)
parsnip model object
##### xgb.Booster
raw: 799.2 Kb
call:
xgboost::xgb.train(params = list(eta = 0.05, max_depth = 5, gamma = 0,
colsample_bytree = 1, colsample_bynode = 0.8, min_child_weight = 1,
subsample = 0.8), data = x$data, nrounds = 2000, watchlist = x$watchlist,
verbose = 0, early_stopping_rounds = 300, eval_metric = "logloss",
nthread = 1, objective = "binary:logistic")
params (as set within xgb.train):
eta = "0.05", max_depth = "5", gamma = "0", colsample_bytree = "1", colsample_bynode = "0.8", min_child_weight = "1", subsample = "0.8", eval_metric = "logloss", nthread = "1", objective = "binary:logistic", validate_parameters = "TRUE"
xgb.attributes:
best_iteration, best_msg, best_ntreelimit, best_score, niter
callbacks:
cb.evaluation.log()
cb.early.stop(stopping_rounds = early_stopping_rounds, maximize = maximize,
verbose = verbose)
# of features: 13
niter: 386
best_iteration : 86
best_ntreelimit : 86
best_score : 0.4452538
best_msg : [86] validation-logloss:0.445254
nfeatures : 13
evaluation_log:
iter validation_logloss
<num> <num>
1 0.6733157
2 0.6546558
--- ---
385 0.4888858
386 0.4891175
学習済のモデルをテストデータに適用し、テストデータに対する分類結果とその正解率を算出します。
<- predict (gbm_fit, d_test)[[1 ]]<- d_test$ Statussprintf ("accuracy : %.4f" , sum (y_pred== y_true)/ length (y_true))
(2) モデル情報の利用
parsnipではextract_fit_engine関数が、そのような機能を提供します。 次の例では、上で学習させたparsnipモデルから、extract_fit_engine関数でxgboostモデルとしての情報を取り出し、xgb.importance関数に渡しています。
<- gbm_fit %>% extract_fit_engine () %>% xgb.importance (model = .)
Feature Gain Cover Frequency
<char> <num> <num> <num>
1: Income 0.162361809 0.15308392 0.15144738
2: Price 0.152862530 0.19189145 0.18225867
3: Amount 0.134619300 0.13369218 0.13318425
4: Seniority 0.118221478 0.10297003 0.08957634
5: Age 0.081321036 0.08125753 0.11454839
6: Records 0.077957485 0.05251855 0.02633868
7: Assets 0.066155159 0.06963897 0.07764940
8: Expenses 0.062748185 0.05693741 0.06994658
9: Job 0.041249101 0.03177091 0.03093552
10: Time 0.036138501 0.03563342 0.04149584
11: Home 0.032315613 0.03410136 0.03640204
12: Debt 0.024199277 0.03237239 0.02932041
13: Marital 0.009850526 0.02413188 0.01689651
この特徴量重要度の情報を利用して、重要度が上位の変数のみを使用した一般化線形モデル(ロジスティック回帰)によるモデルを構築することを考えます。 特徴量重要度(Gain)の数値が、ある一定の値を超えている変数のみを使用して、parsnipを通したロジスティック回帰モデルを作成します。 ロジスティック回帰はlogistic_reg関数を使用しますが、モデル構築から予測までの流れはやはり、ランダムフォレストやGBMと同じです。
<- na.omit (credit_data)<- d3[1 : n_train,]<- d3[(n_train+ 1 ): nrow (d3),]<- logistic_reg () %>% set_mode ("classification" ) %>% set_engine ("glm" ) %>% # エンジンとしてR標準のglmを使用 fit_xy (x= d_train[, importance_gbm[importance_gbm$ Gain > 0.05 ,][["Feature" ]]], y= d_train$ Status)
parsnip model object
Call: stats::glm(formula = ..y ~ ., family = stats::binomial, data = data)
Coefficients:
(Intercept) Income Price Amount Seniority Age
1.094e+00 7.647e-03 1.310e-03 -2.288e-03 1.075e-01 -6.543e-03
Recordsyes Assets Expenses
-1.846e+00 2.478e-05 -1.219e-02
Degrees of Freedom: 3028 Total (i.e. Null); 3020 Residual
Null Deviance: 3410
Residual Deviance: 2683 AIC: 2701
<- predict (glm_fit, d_test)[[1 ]]<- d_test$ Statussprintf ("accuracy : %.4f" , sum (y_pred== y_true)/ length (y_true))
ここではロジスティック回帰のエンジンとしてR標準のglmを使用していますが、glmのモデルは、summary関数で各説明変数の標準誤差等のより詳細な情報が確認できます。 そこで再度extract_fit_engine関数を使用して、parsnipモデルからglmモデルとしての情報を取り出し、summary関数に渡します。
%>% extract_fit_engine () %>% summary (.)
Call:
stats::glm(formula = ..y ~ ., family = stats::binomial, data = data)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 1.094e+00 2.274e-01 4.811 1.50e-06 ***
Income 7.647e-03 8.305e-04 9.207 < 2e-16 ***
Price 1.310e-03 1.634e-04 8.021 1.05e-15 ***
Amount -2.288e-03 1.966e-04 -11.641 < 2e-16 ***
Seniority 1.075e-01 9.039e-03 11.890 < 2e-16 ***
Age -6.543e-03 5.264e-03 -1.243 0.21388
Recordsyes -1.846e+00 1.287e-01 -14.340 < 2e-16 ***
Assets 2.478e-05 7.969e-06 3.109 0.00188 **
Expenses -1.219e-02 2.582e-03 -4.722 2.33e-06 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 3410.5 on 3028 degrees of freedom
Residual deviance: 2682.8 on 3020 degrees of freedom
AIC: 2700.8
Number of Fisher Scoring iterations: 5
parsnipで様々なモデルを使用する
ここまでの例で示してきたように、parsnipではモデルの種類、エンジン、モードの組み合わせでモデルを特定します。 あるモデルの種類に関して、parsnipが対応しているパッケージ(エンジン)とモードの組み合わせを表示するshow_engines関数が用意されています。 例えばGBMについてこれらの情報を知りたい場合、show_engines関数に、GBMの生成関数の名称である”boost_tree”を引数として渡して実行します。
show_engines ("boost_tree" )
# A tibble: 5 × 2
engine mode
<chr> <chr>
1 xgboost classification
2 xgboost regression
3 C5.0 classification
4 spark classification
5 spark regression
tidymodelsの公式サイトではparsnipが対応しているモデル、エンジン、モードの全ての組み合わせが一覧化されているので、そちらもご参照ください。
また、更に発展的な使用方法として、parsnipに用意されていないモデルを新たに登録し、parsnipのインターフェースで実行できるようにする方法も用意されています。 これにより、parsnipベースで記述したコードを再利用できたり、tidymodelsの他のパッケージとの連携が可能になるといった利点が考えられます。 モデルの追加はparsnipのディベロッパー・ツールとして用意されている関数を使用します。 次のコードは新たなモデルとそのモード、エンジンとなるパッケージを登録しています。
# 架空のnewpkgパッケージをnew_modelの回帰モデルとして登録する set_new_model ("new_model" )set_model_mode (model = "new_model" , mode = "regression" )set_model_engine (model = "new_model" ,mode = "regression" ,eng = "newpkg" set_dependency ("new_model" , eng = "newpkg" , pkg = "newpkg" )show_model_info ("new_model" )
Information for `new_model`
modes: unknown, regression
engines:
regression: newpkgNA
no registered arguments.
no registered fit modules.
no registered prediction modules.
実際に使用するには更に、モデル生成関数の作成、引数の設定、fit関数及びpreditct関数に対する動作の設定等が必要になります。 内容はやや高度になりますので、興味がある方はtidymodels公式サイトの以下記事をご参照ください。