パッケージの概要
機械学習におけるRandomForestモデルを構築できます。高速実装であり、特に高次元データに適しています。 分類木、回帰木、生存木、確率予測木のアンサンブルをサポートしています。
使用例:irisデータの分類
irisデータを用いて、がく弁・花弁の長さ・幅の情報からアヤメの種類を特定するRandomForestモデルをrangerパッケージを用いて構築します。
irisデータセットを読み込む
irisデータを読み込み、データの先頭を表示します。
- Sepal.Length:がく弁の長さ
- Sepal.Width:がく弁の幅
- Petal.Length:花弁の長さ
- Petal.Width:花弁の幅
アヤメの種類はsetosa(1)、versicolor(2)、virginica(3)の3種類です。
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
irisデータの構造
irisデータの各種構造を確認します。
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
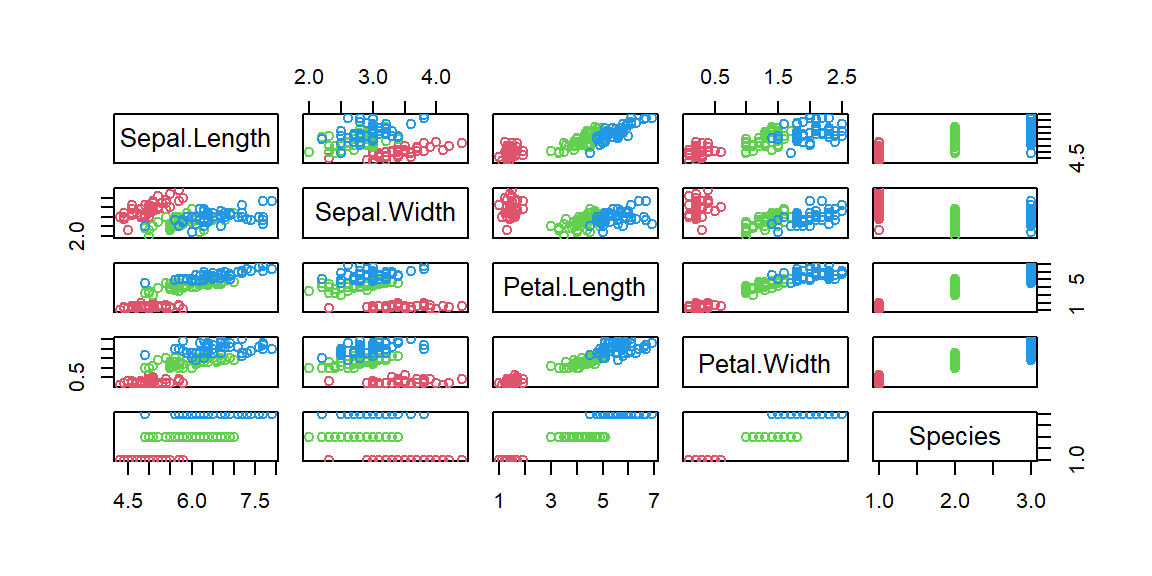
また、データを散布図にプロットして確認します。
plot(iris, col=c(2, 3, 4)[iris$Species])
モデル構築1(全体データ)
まずは全てのデータを使ってRandomForestモデルを構築してみます。
library(ranger)
# シードを設定
set.seed(123)
(model.all <- ranger(Species ~ ., data = iris, importance = "impurity"))
Ranger result
Call:
ranger(Species ~ ., data = iris, importance = "impurity")
Type: Classification
Number of trees: 500
Sample size: 150
Number of independent variables: 4
Mtry: 2
Target node size: 1
Variable importance mode: impurity
Splitrule: gini
OOB prediction error: 5.33 %
分類木の構築においては、importance = “impurity”と設定することにより、結果にvariable.importanceを保持してくれます。この中身を確認することにより各変数の重要度を確認することが出来ます。irisデータの分類には花弁の長さ(Petal.Length)・花弁の幅(Petal.Width)の情報が重要であることが分かります。
model.all$variable.importance
Sepal.Length Sepal.Width Petal.Length Petal.Width
9.629622 2.453766 41.714390 45.460729
データセットの準備
irisデータをモデル生成のための訓練データと、モデル評価のためのテストデータに分割します。データ割合は訓練データを7割、テストデータを3割とします。確認のため、データサイズを出力します。
# 再現性のためにシードを設定
set.seed(123)
# データの分割
sample_indices <- sample(1:nrow(iris), 0.7 * nrow(iris))
train_data <- iris[sample_indices, ]
test_data <- iris[-sample_indices, ]
# データサイズの確認
c(nrow(iris), nrow(train_data), nrow(test_data))
モデルの生成・予測の実行
訓練データを用いて分類木のモデルを生成します。モデルの生成結果は以下の通りです。
set.seed(123)
(model <- ranger(Species ~ ., data = train_data))
Ranger result
Call:
ranger(Species ~ ., data = train_data)
Type: Classification
Number of trees: 500
Sample size: 105
Number of independent variables: 4
Mtry: 2
Target node size: 1
Variable importance mode: none
Splitrule: gini
OOB prediction error: 5.71 %
テストデータを用いてモデルの評価をします。まずは、テストデータを先ほど構築した分類木モデルに適用させ、その予測結果をpredictionsに格納します。
predictions <- predict(model, data = test_data)$predictions
予測結果とテストデータのもともとのアヤメの分類とを比較します。おおむね正しく分類できていることが分かります。
(confusion_matrix <- table(predictions, test_data$Species))
predictions setosa versicolor virginica
setosa 14 0 0
versicolor 0 17 0
virginica 0 1 13
ハイパーパラメーターのチューニング
rangerのRandomForestモデルにおける主なハイパーパラメーターは以下の通りです。
- 決定木を生成する際に使用するパラメータの数(mtry)
- 生成する決定木の数(num.trees)
これらのハイパーパラメーターの最適な設定を探す作業がハイパーパラメーターのチューニングとなります。rangerのハイパーパラメーターのチューニング用にはtuneRanger等のパッケージがありますが、ここではnum.treesについて直接パラメータ設定を変更して精度比較を実施します。
なお、rangerのRandomForestモデルではOOBError(Out-Of-bag Error)が算出されます。これはモデル構築時に一部データを学習に使用しない代わりにモデル検証に使用して誤差率を求めています。そのため、クロスバリデーションをしなくても、ある程度の汎化性能を測ることができます。
num.trees = 300としてモデル構築します。OOBErrorは5.71%です。
set.seed(123)
(model.num.trees.300 <- ranger(Species ~ ., data = train_data, num.trees = 300))
Ranger result
Call:
ranger(Species ~ ., data = train_data, num.trees = 300)
Type: Classification
Number of trees: 300
Sample size: 105
Number of independent variables: 4
Mtry: 2
Target node size: 1
Variable importance mode: none
Splitrule: gini
OOB prediction error: 5.71 %
num.trees = 500としてモデル構築します。OOBErrorは5.71%です。
set.seed(123)
(model.num.trees.500 <- ranger(Species ~ ., data = train_data, num.trees = 500))
Ranger result
Call:
ranger(Species ~ ., data = train_data, num.trees = 500)
Type: Classification
Number of trees: 500
Sample size: 105
Number of independent variables: 4
Mtry: 2
Target node size: 1
Variable importance mode: none
Splitrule: gini
OOB prediction error: 5.71 %
num.trees = 700としてモデル構築します。OOBErrorは4.76%です。
set.seed(123)
(model.num.trees.700 <- ranger(Species ~ ., data = train_data, num.trees = 700))
Ranger result
Call:
ranger(Species ~ ., data = train_data, num.trees = 700)
Type: Classification
Number of trees: 700
Sample size: 105
Number of independent variables: 4
Mtry: 2
Target node size: 1
Variable importance mode: none
Splitrule: gini
OOB prediction error: 4.76 %
num.trees = 700のときにOOBErrorが最も小さくなったので、そのモデルにてテストデータで精度を測ってみます。もともと精度が高いため、結果は変わりませんでした。
predictions.num.trees <- predict(model.num.trees.700, data = test_data)$predictions
(confusion_matrix <- table(predictions.num.trees, test_data$Species))
predictions.num.trees setosa versicolor virginica
setosa 14 0 0
versicolor 0 17 0
virginica 0 1 13