パッケージの概要
機械学習におけるRandomForestモデルの構築を行えます。また、tuneRangerではハイパーパラメータのチューニングを行う機能も提供されます。
使用例:irisデータの分類
irisデータを用いて、がく弁・花弁の長さ・幅の情報からアヤメの種類を特定するRandomForestモデルをtuneRangerパッケージを用いて構築します。
irisデータセットを読み込む
irisデータを読み込み、データの先頭を表示します。
- Sepal.Length:がく弁の長さ
- Sepal.Width:がく弁の幅
- Petal.Length:花弁の長さ
- Petal.Width:花弁の幅
アヤメの種類はsetosa(1)、versicolor(2)、virginica(3)の3種類です
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
irisデータの構造
irisデータの各種構造を確認します。
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
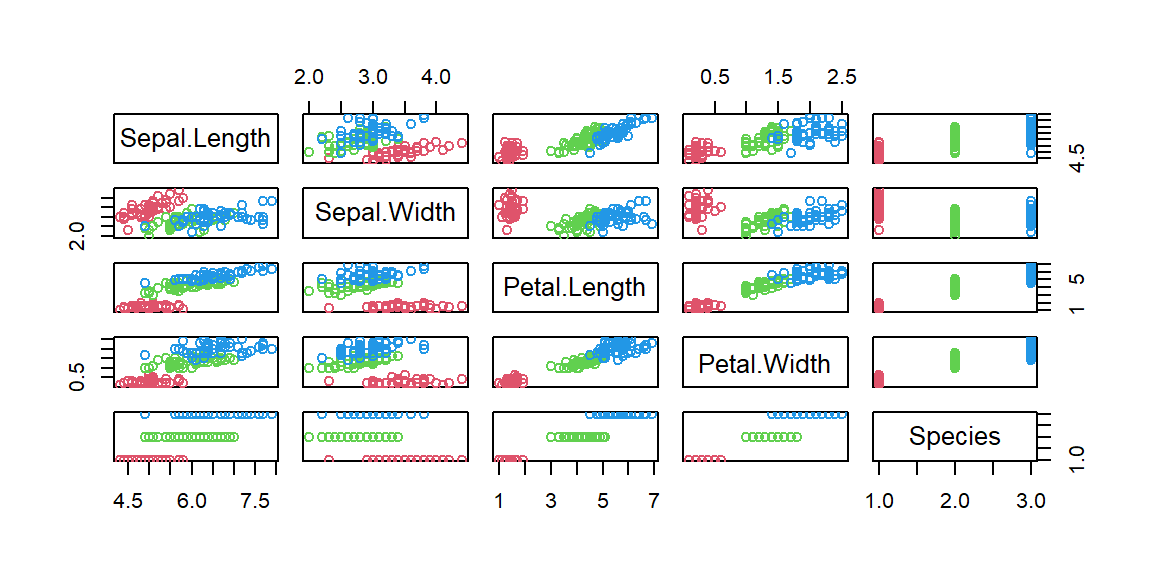
また、データを散布図にプロットして確認します。
plot(iris, col=c(2, 3, 4)[iris$Species])
データセットの準備
irisデータをモデル生成のための訓練データと、モデル評価のためのテストデータに分割します。データ割合は訓練データを7割、テストデータを3割とします
# 再現性のためにシードを設定
set.seed(100)
# データの分割
n <- nrow(iris)
train.rate <- 0.7
# データそのものではなく、データ番号を吐き出している
(train.set <- sample(n, n * train.rate))
[1] 102 112 4 55 70 98 135 7 43 140 51 25 2 68 137 48 32 85
[19] 91 121 16 116 66 146 93 45 30 124 126 87 95 97 120 29 92 31
[37] 54 41 105 113 24 142 143 63 65 9 150 20 14 78 88 3 36 27
[55] 46 59 96 69 47 147 129 136 12 141 130 56 22 82 53 99 5 44
[73] 28 52 139 42 15 57 75 37 26 110 100 149 132 107 35 58 127 111
[91] 144 86 114 71 123 119 18 8 128 83 138 19 115 23 89
(test.set <- setdiff(1:n, train.set))
[1] 1 6 10 11 13 17 21 33 34 38 39 40 49 50 60 61 62 64 67
[20] 72 73 74 76 77 79 80 81 84 90 94 101 103 104 106 108 109 117 118
[39] 122 125 131 133 134 145 148
モデル生成
makeClassifTaskにてタスクの定義を行い、makeLeanerにて適用するアルゴリズムの選択を行います。
(task <- makeClassifTask(data = iris, target = "Species"))
Supervised task: iris
Type: classif
Target: Species
Observations: 150
Features:
numerics factors ordered functionals
4 0 0 0
Missings: FALSE
Has weights: FALSE
Has blocking: FALSE
Has coordinates: FALSE
Classes: 3
setosa versicolor virginica
50 50 50
Positive class: NA
lrn <- makeLearner("classif.lda")
分割したデータセットを基にモデルの訓練を行います。また、訓練時の誤分類率(mmce)や精度(ace)を把握することが出来ます。
(model <- train(lrn, task, subset = train.set))
Model for learner.id=classif.lda; learner.class=classif.lda
Trained on: task.id = iris; obs = 105; features = 4
Hyperparameters:
パラメータチューニング
パラメータチューニングのための実行時間を事前に知ることが出来ます。
estimateTimeTuneRanger(task, num.trees = 500, num.threads = 3, iters = 30)
Approximated time for tuning: 50S
tuneRangerを使用して、実際にパラメータチューニングをやってみます。
チューニング後のモデルの精度を確認してみます。精度が向上していることが分かります。
pred.res <- predict(res$model, task = task, subset = test.set)
performance(pred.res, measures = list(mmce, acc))
mmce acc
0.02222222 0.97777778