パッケージの概要
rpartは再帰的分割による回帰木・分類木の実装を与えます。また、rpart.plotで決定木の可視化が可能です。
使用例:irisデータの分類
irisデータを用いて、がく弁・花弁の長さ・幅の情報からアヤメの種類を特定する分類モデルを作成します。
irisデータセットを読み込む
irisデータを読み込み、データの先頭を表示します。
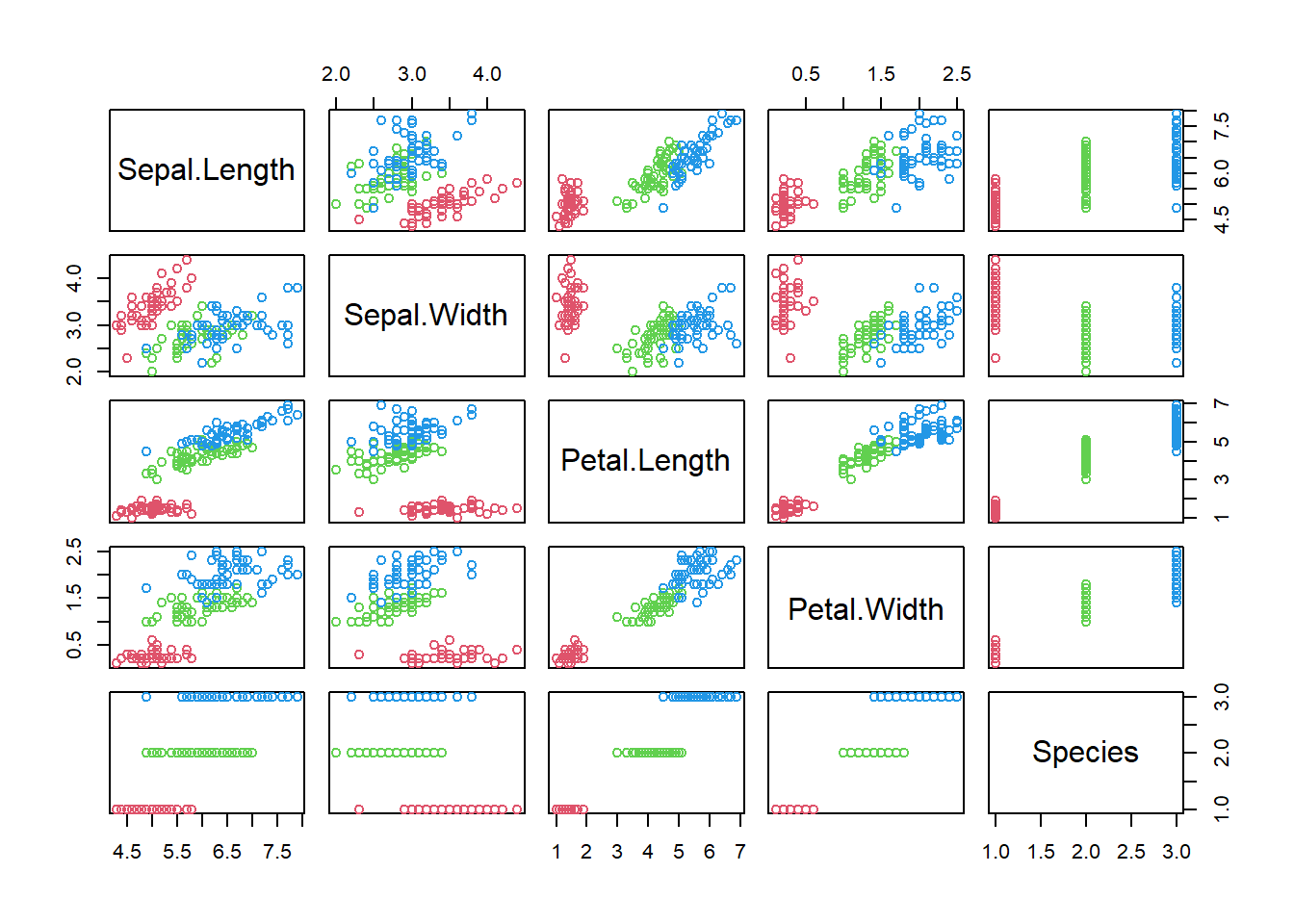
- Sepal.Length:がく弁の長さ
- Sepal.Width:がく弁の幅
- Petal.Length:花弁の長さ
- Petal.Width:花弁の幅
アヤメの種類はsetosa(1)、versicolor(2)、virginica(3)の3種類です。
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
irisデータの構造
irisデータの各種構造を確認します。
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
また、データを散布図にプロットして確認します。
plot(iris, col=c(2, 3, 4)[iris$Species])
モデル構築1(全体データ)
まずは全てのデータを使って分類木モデルを構築してみます。
library(rpart)
library(rpart.plot)
Warning: package 'rpart.plot' was built under R version 4.5.1
# シードを設定
set.seed(123)
(iris.rp <- rpart(Species ~ ., data = iris))
n= 150
node), split, n, loss, yval, (yprob)
* denotes terminal node
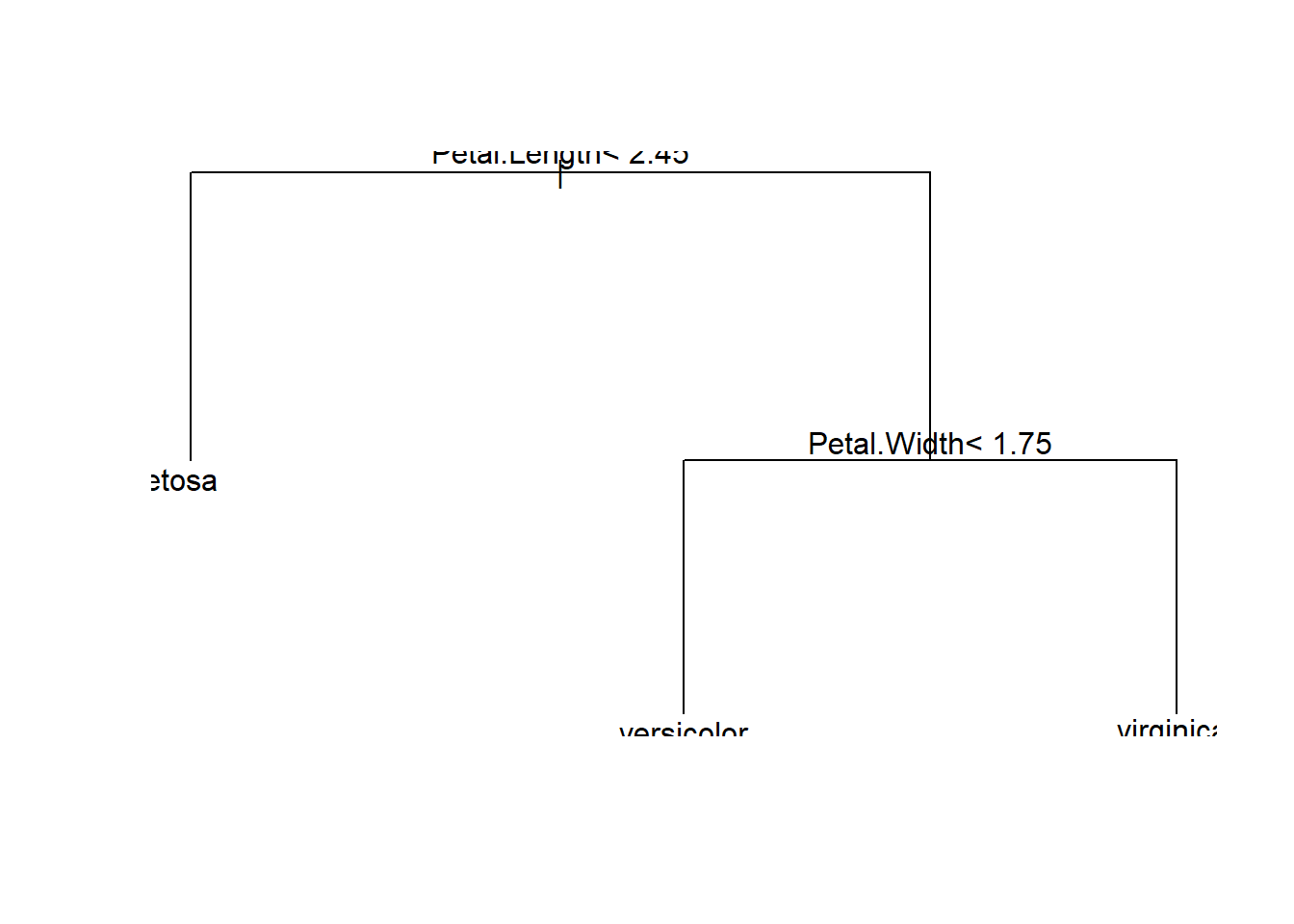
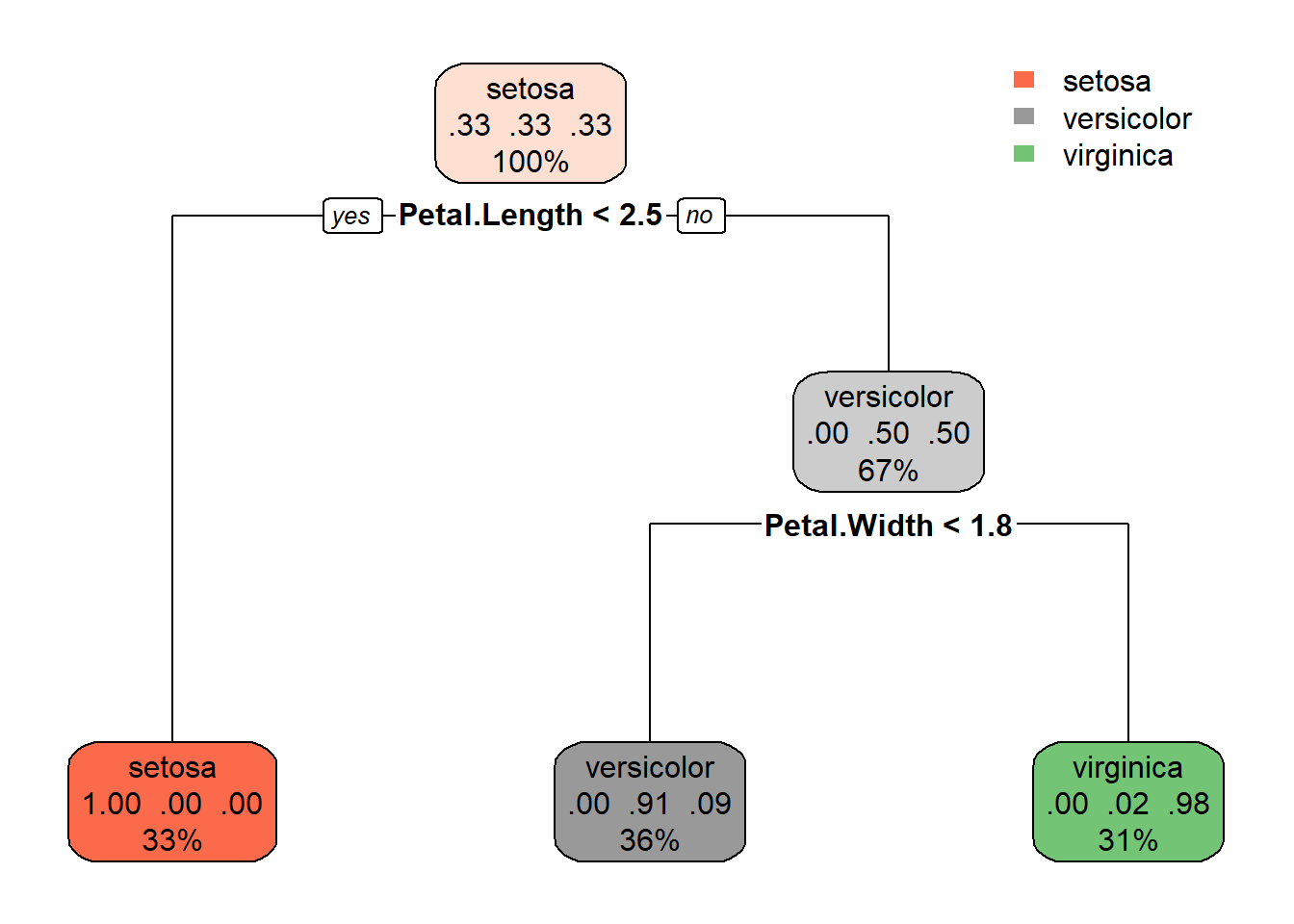
1) root 150 100 setosa (0.33333333 0.33333333 0.33333333)
2) Petal.Length< 2.45 50 0 setosa (1.00000000 0.00000000 0.00000000) *
3) Petal.Length>=2.45 100 50 versicolor (0.00000000 0.50000000 0.50000000)
6) Petal.Width< 1.75 54 5 versicolor (0.00000000 0.90740741 0.09259259) *
7) Petal.Width>=1.75 46 1 virginica (0.00000000 0.02173913 0.97826087) *
可視化
モデルを可視化します。plotで木の構造(分岐)を表示し、textで各ノードの分岐の基準や分類ラベルを表示します。デフォルトの設定だと図が見切れてしまうことがあります。
plot(iris.rp)
text(iris.rp)
rpart.plotを用いると、分類木をより分かりやすく表示させることができます。
モデル構築2(訓練データとテストデータに分割)
irisデータをモデル生成のための訓練データと、モデル評価のためのテストデータに分割します。データ割合は訓練データを7割、テストデータを3割とします。確認のため、データサイズを出力します。
# シードを設定
set.seed(123)
# データの分割
sample_indices <- sample(1:nrow(iris), 0.7 * nrow(iris))
df.train <- iris[sample_indices, ]
df.test <- iris[-sample_indices, ]
# データサイズの確認
c(nrow(iris), nrow(df.train), nrow(df.test))
モデル生成
訓練データを用いて分類木モデルを生成します。
# シードを設定
set.seed(123)
(model.rp <- rpart(Species ~ ., data = df.train))
n= 105
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 105 68 virginica (0.34285714 0.30476190 0.35238095)
2) Petal.Length< 2.45 36 0 setosa (1.00000000 0.00000000 0.00000000) *
3) Petal.Length>=2.45 69 32 virginica (0.00000000 0.46376812 0.53623188)
6) Petal.Width< 1.75 35 4 versicolor (0.00000000 0.88571429 0.11428571) *
7) Petal.Width>=1.75 34 1 virginica (0.00000000 0.02941176 0.97058824) *
モデル評価
テストデータを使ってモデル評価を行います。まずはテストデータを元に生成したモデルを用いて予測結果を算出します。
prediction <- predict(model.rp, df.test, type = "class")
予測結果とテストデータのもともとのアヤメの分類とを比較します。おおむね正しく分類できていることが分かります。
(result <- table(prediction, df.test$Species))
prediction setosa versicolor virginica
setosa 14 0 0
versicolor 0 18 1
virginica 0 0 12
(accuracy_prediction <- sum(diag(result)) / sum(result))
ハイパーパラメーターのチューニング
rpartの主なハイパーパラメーターは以下の通りです。
- 木の複雑度に関するパラメータ(cp)
- ノード分割の最小サンプル数(minsplit)
- 木の最大の深さ(maxdepth)
これらのハイパーパラメータの最適な設定を探す作業がハイパーパラメーターのチューニングとなります。
まずはcpの最適な設定を確認します。これはprintcpを用いることができます。
Classification tree:
rpart(formula = Species ~ ., data = df.train)
Variables actually used in tree construction:
[1] Petal.Length Petal.Width
Root node error: 68/105 = 0.64762
n= 105
CP nsplit rel error xerror xstd
1 0.52941 0 1.000000 1.10294 0.068075
2 0.39706 1 0.470588 0.47059 0.069364
3 0.01000 2 0.073529 0.11765 0.039979
xerror(交差検証誤差)が最も低くなるcpは0.01でした。これはデフォルトの設定と一致します。
次に、minsplitのチューニングを行います。簡便的にテストデータでの設定の差を確認します。なお、デフォルトの設定は20です。
# シードを設定
set.seed(123)
# 候補となる minsplit の値
minsplit_values <- c(5, 20, 40)
# minsplit ごとの精度を格納するデータフレーム
results <- data.frame(minsplit = minsplit_values, Accuracy = NA)
# 各 minsplit のモデルを作成し、精度を測定
for (i in seq_along(minsplit_values)) {
control <- rpart.control(minsplit = minsplit_values[i])
model <- rpart(Species ~ ., data = df.train, method = "class", control = control)
# 予測
predictions <- predict(model, df.test, type = "class")
accuracy <- mean(predictions == df.test$Species)
# 結果を保存
results$Accuracy[i] <- accuracy
}
# 結果の確認
print(results)
minsplit Accuracy
1 5 0.9777778
2 20 0.9777778
3 40 0.9777778
irisデータだと特段変化がないようです。
最後にmaxdepthについても同様に試してみます。デフォルトの設定は5です。
# シードを設定
set.seed(123)
# 候補となる maxdepth の値
maxdepth_values <- c(3, 5, 7)
# maxdepth ごとの精度を格納するデータフレーム
results <- data.frame(maxdepth = maxdepth_values, Accuracy = NA)
# 各 minsplit のモデルを作成し、精度を測定
for (i in seq_along(minsplit_values)) {
control <- rpart.control(maxdepth = maxdepth_values[i])
model <- rpart(Species ~ ., data = df.train, method = "class", control = control)
# 予測
predictions <- predict(model, df.test, type = "class")
accuracy <- mean(predictions == df.test$Species)
# 結果を保存
results$Accuracy[i] <- accuracy
}
# 結果の確認
print(results)
maxdepth Accuracy
1 3 0.9777778
2 5 0.9777778
3 7 0.9777778
こちらもirisデータだとと特段変化がないようです。